Mírenlo bien: esta estructura es la proteína S del COVID19 y puede ser la molécula que nos permita encontrar un fármaco que pueda tratar al COVID19.
La actual pandemia del beta coronavirus de Wuhan ( COVID-19) está mediada por un virus RNA monocatenario, de tamaño 50-200 nanometros, con una serie de azúcares en la cubierta externa lo que le hace ser "relativamente pesado". Parece ser que la vida media del virus en aerosol es de 66 minutos, y que al cabod e las tres horas se deporita en el suelo. Esto es muy arreisgado y las declaraciones de una portavoz de la OMS , la Dra Maria van Kerkhouve diciendo que no hace falta mascarillas porque no es una transmisión "airborne" nos parecen UNA TEMERIDAD.
El virus COVIS-19 es MUY contagioso con una R0 = 2,7, muy superior a los ortomixovirus de la "gripe vulgar" que es de R0=1,3.
Aunque les cueste creerlo, se puede patentar la secuencia de ARN de un virus, que aunque parezca natural ( o no, y es una arma biolñógica) no deja de ser un sisnsentido patentarlo como si fuera un dentífrico. Los virus SARS y MERS fueron patentados, como atestigua:
https://patentimages.storage.googleapis.com/6b/c3/21/a62eb55a0e678c/US7220852.pdf y MERS-CoV (MERS) https://data.epo.org/publication-server/pdf-document?pn=2898067&ki=B1&cc=EP&pd=20200115
En sólamente dos meses el COVID-19 está siendo exhaustivamente estudiado: por la encuesta epidemiológica se supone que es infectante en la fase asintomática ( de 2 a 14 días, con una media de 5 días) y también en las dos semanas posteriores a la "curación· de la enfermedad.
En la superficie del virus nos encontramos las proteínas S ( con dos subunidades S1 y S2) , y la proteína M .
El ARN monocatenario del COVID-19 codifica la poliproteína
ORF1b que se escinde en varias proteínas relevantes en su replicación.
Una de ellas es la polimerasa de ARN dependiente de ARN, llamada RdRp, o nsp12, clave en la catálisis de la síntesis del ARN del virus y diana de fármacos antivirales como remdesivir y sofosbuvir.
Se acaba de publicar en bioRxiv la estructura tridimensional del complejo proteico nsp12-nsp7-nsp8; se ha obtenido por criomicroscopia electrónica (cryo-EM) con una resolución de 2.9 Å. Además de ayudar a entender cómo actúan remdesivir y sofosbuvir, se espera que permita desarrollar nuevos antivirales específicos
Yan Gao, Liming Yan, …, Zihe Rao, «Structure of RNA-dependent RNA polymerase from 2019-nCoV, a major antiviral drug target,» bioRxiv preprint (16 Mar 2020), doi: https://doi.org/10.1101/2020.03.16.993386. Seguro que se publicará en Nature Communications, como ya lo hizo el artículo sobre SARS-CoV, Robert N. Kirchdoerfer, Andrew B. Ward, «Structure of the SARS-CoV nsp12 polymerase bound to nsp7 and nsp8 co-factors,» Nature Communications 10: 2342 (28 May 2019), doi: https://doi.org/10.1038/s41467-019-10280-3.

La estructura 3D del complejo proteico nsp12-nsp7-nsp8 del coronavirus humano SARS-CoV se publicó en Nature Communications en 2019. Comparte muchas semejanzas (estructuras conservadas) con la de SARS-CoV-2 (antes llamado 2019-nCoV). Pero las pocas diferencias son claves para el desarrollo de antivirales específicos contra COVID-19. Esta figura muestra las diferencias entre las secuencias de aminoácidos de la proteína nsp12 del nuevo coronavirus humano SARS-CoV-2 (2019-nCoV en la figura), el coronavirus humano SARS-CoV y el coronavirus de murciélagos RaTG13. Parecen pocas diferencias, pero son suficientes para que los antivirales más eficaces contra SARS no sean los más eficaces contra COVID-19.
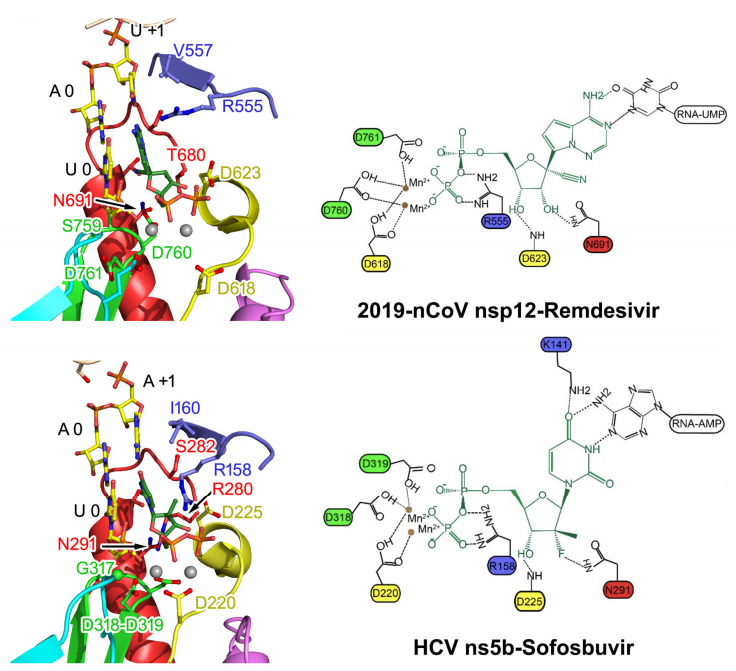
La estructura tridimensional a escala atómica de la proteína nsp12 permite estudiar con todo lujo de detalles su interacción con antivirales.
El fármaco que más posibilidades tiene de ser útil es el Remdesivir, del laboratorio americano Gilead, que se va a estudiar en el Ensayo Clínico "Solidaruty" de la OMS.s
Fuentes: (Manli Wang, Ruiyuan Cao, …, Gengfu Xiao, «Remdesivir and chloroquine effectively inhibit the recently emerged novel coronavirus (2019-nCoV) in vitro,» Cell Research 30: 269-271 (04 Feb 2020), doi: https://doi.org/10.1038/s41422-020-0282-0; Michelle L. Holshue, Chas DeBolt, …, Satish K. Pillai, «First Case of 2019 Novel Coronavirus in the United States,» The New England Journal of Medicine (NEJM) 382:929-936 (05 Mar 2020), doi: https://doi.org/10.1056/NEJMoa2001191).
y tres tipos de proteínas, la proteína Espikulada ( Spike proteín) ,que es tiene una estructura conformacional representada en el video que tienen abajo.
Esta proteína S está compuesta por dos subunidades , S1:encargada del ANCLAJE del virus;
y S2, que es la que se encarga de la FUSION y posterior introducción a la célula del pulmón, uniéndose al receptor ACE/2..
ésta proteína S ha sido secuenciada y su estructura está mostrada abajo:
formando parte del genoma del virus y tiene entre 1120 y 1200 kdalton.
En la zona de líneas discontínuas de abajo
Es la forma mediante la cual el virus se une a un receptor ACE/2 ( angiotensina y el receptor RBD del COVID-19.
Este receptor es más abundante en las células epiteliales de pulmón y , que vemos estructuralmente aquí abajo por microscopía crioelectrónica. Esta técnica fue premiada con el premio Nobeld e medicina, hace dos años.:
Si ampliamos un poco más la imagen , vemos como ése receptor va a ser menos abundante en niños, por debajod e 16 años, quizás por eso es por lo que hay menos casos en infantes. Al parecer sólo ha habido muy pcos casos y quizás ea porque en esas edades tempranas de la vida el sistema ACe/2 de la angiotensina está muy inmduro. Hay pocos niños afortunadamente con hipertensión renal por exceso de angiotensina.
O más aumentada la imagen:
Otra de las estrategias de ataque sería la presencia de una MPro, o proteasa que está igualmente en la superficie del virus y tiene ésta estructura:
Esta proteasa se ha visto que es inhibida por una serie de sustancias que se llaman AKG, inhibidores del alfa Ceto glutamida, que son pro fármacos activados por peróxidod e hidrógeno, con la estructura básica:
cuya estructura química tenemos abajo: son los compuestos experimentales 13 a,13 b y 11 r.
Estructura química de los inhibidores de la alfa cetoglutamida ( AKG) , denominados compuesto 11r, 13 a y 13 b.
Estas sustancias inhiben la proteasa del COVID-19.tal como queda reflejado en l a siguiente imagen, que es la unión específica al compuesto 13 b.:se produce un ataque nucleofílico de la cys en el grupo cetónico y se forma un tio hemi ketal, en reacción reversible, lo que se sabe por la densidad electrónica de ésta molécula quiral
El grupo oxidrilo de éste tio hemi ketal se estabiliza por enlaces de puentes de hidrógeno desde la cadena de amidas de los aminoácidos Cisteína y Glicina (y parcialmente serina)
el RESTO DE LA EXPLICACIÓN es un manual de química farmacéutica que es una delicia no truducir y leerlo directamente del ingles:
Compound 13b inhibits the purified recombinant 2019-nCoV Mpro with IC50 = 0.67 ± 0.18 μM. The corresponding IC50 values for inhibition of the SARS-CoV Mpro and the MERS-CoV Mpro are 0.9 ± 0.29 μM and 0.58 ± 0.22 μM, respectively. In a SARS-CoV replicon (Kusov et al., 2015), RNA replication is inhibited with EC50 = 1.75 ± 0.25 μM. In human Calu3 cells infected with MERS-CoV, the compound showed excellent antiviral activity (Lucie Sauerhering & Stephan Becker personal communication).
As the next steps in the development of compound 13b towards a potential drug targeting 2019-nCoV or other coronaviruses, we will undertake tests in virus-infected cell cultures and in a small-animal model (once it will become available for 2019-nCoV). Meanwhile, our crystal structures may be used by others for virtual screening and de-novo design of inhibitors.
















No hay comentarios:
Publicar un comentario